微生物基因组的de novo测序(De novo sequencing),不依赖于参考基因组信息,利用生物信息学分析方法对目标菌株的测序数据进行基因组的从头组装(De novo assembly),获得菌株的基因组序列。基于组装结果,结合数据库对基因组进行注释,并以此为基础开展下游分析。
本技术路线适合基因组中存在大片段基因插入/缺失,缺少近缘参考基因组的菌种,如肺炎克雷伯(*Klebsiella pneumoniae*)、无乳链球菌(*Streptococcus agalactiae*)等。通过de novo测序的分析方法,对基因组进行组装,可以对基因组的结构和功能进行精细、深入地研究。
测序数据质控
测序得到的原始数据会存在一定比例的低质量数据,为了保证后续信息分析结果的准确可靠,首先要对原始数据进行过滤处理,得到有效数据。
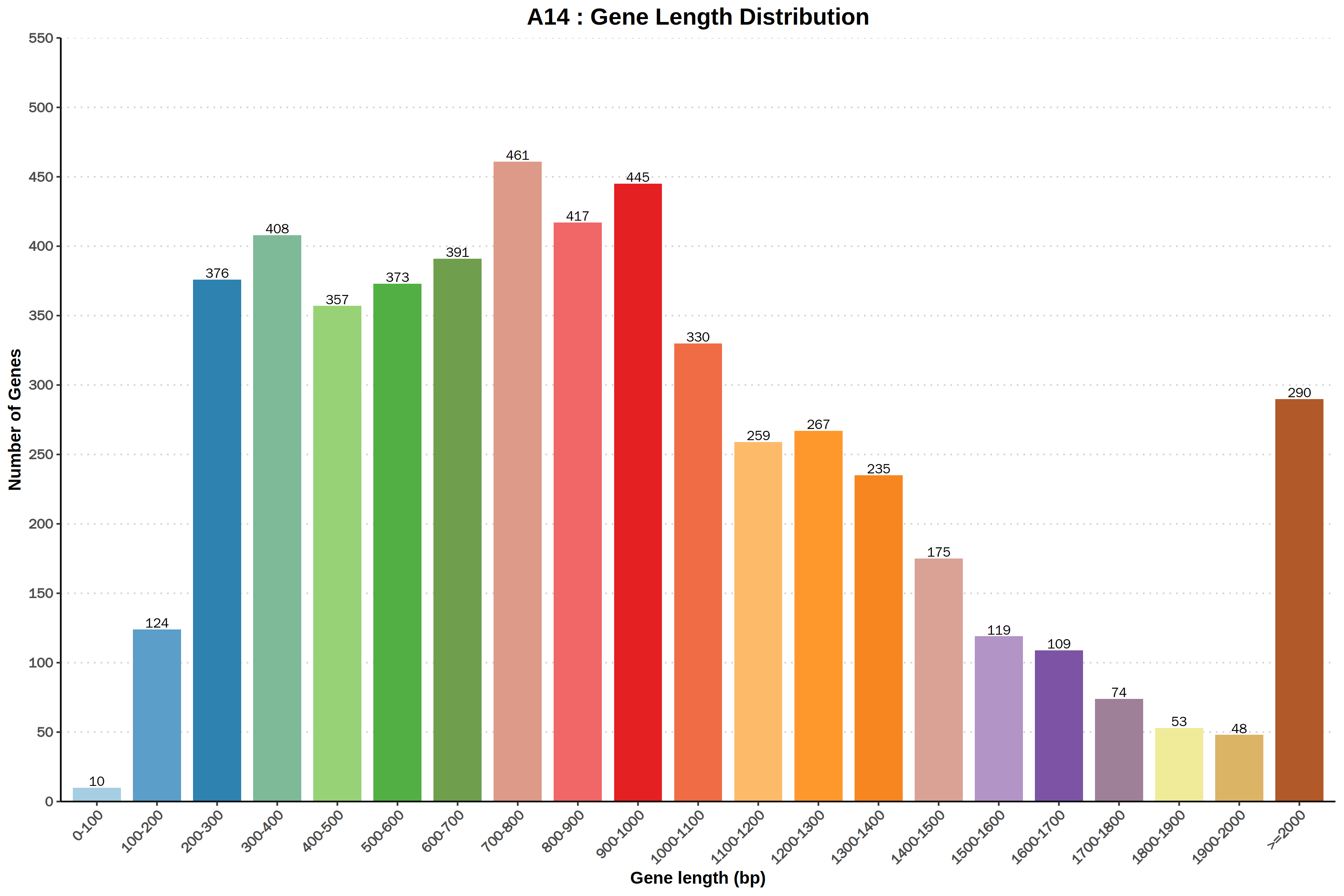
基因组组分分析
微生物基因组包含的功能区域非常丰富,除编码基因区域,更有非编码区域实现转录调控、转录后调控、翻译调控、表观遗传调控等功能,部分功能区域还与物种进化的多样性存在关系。使用Prokka软件对新测序的基因组进行编码基因预测。
基因组功能注释
通过一系列基本功能数据库的注释,获得编码基因在不同数据库中的同源信息,以解释编码基因的生理功能。以KEGG、GO、COG为例,可以在将基因的功能归属到丰富的功能分类中去。
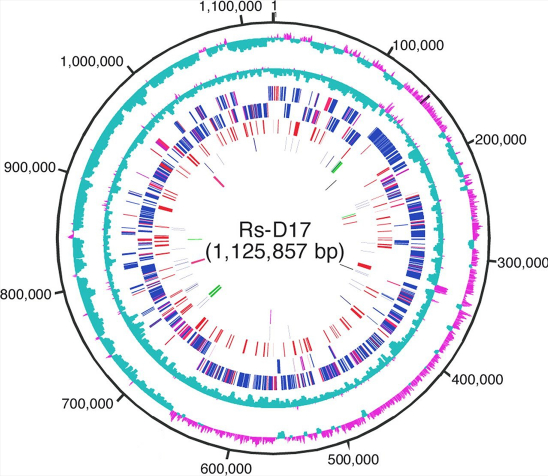
基因组结构总览
用于展示全基因组范围的基因分布、KEGG、GO、COG等基本基因功能分布、ncRNA分布、基因组GC含量及GC-skew值分布,可以提供基因组整体信息的总览和概括。
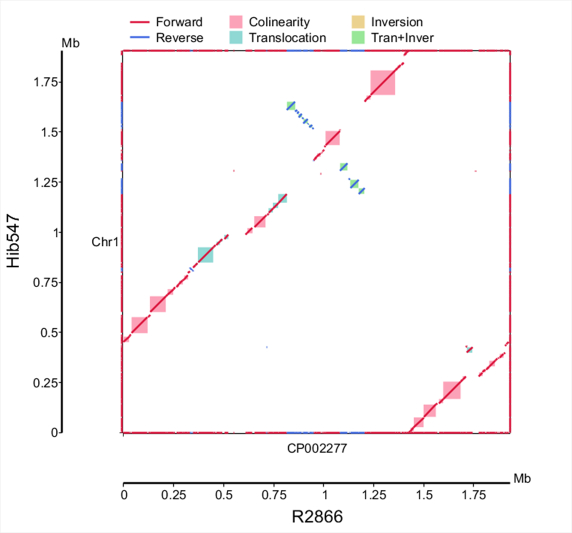
比较基因组研究
通过软件比对分析,可以解析不同基因组之间的共线性关系。
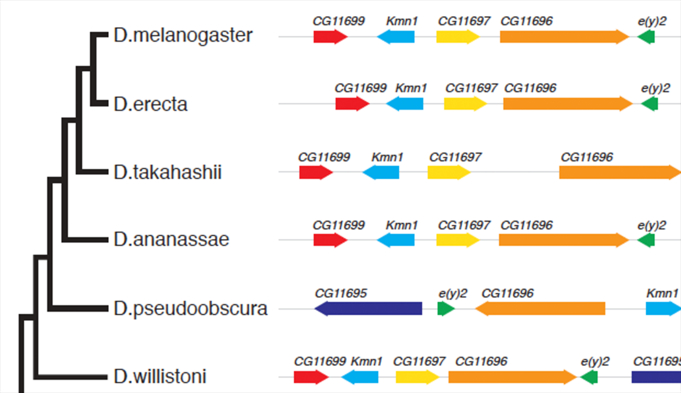
基因组进化分析
基于多个基因组间的单拷贝基因信息,可以构建菌株间的系统进化树,用于解析菌株之间的进化关系。